ISSN 2410-5708 / e-ISSN 2313-7215
Año 9 | Núm. 24 | pág. 45-57 | febrero – mayo 2020
© Copyright (2020). Universidad Nacional Autónoma de Nicaragua, Managua.
Esta obra está bajo una licencia Creative Commons
Atribución-NoComercial-SinDerivar 4.0 Internacional
Evaluación de metodologías de extracción de ADN de plantas recalcitrantes
https://doi.org/10.5377/torreon.v9i24.9723
Fecha de recepción: 13 de febrero, 2020 / Fecha de aceptación: 20 de febrero, 2020
M.Sc. Samantha Miranda Calero
Investigadora
Laboratorio de Biotecnología, UNAN-Managua
Ing. Ena Rivers Carcache
Investigadora
Laboratorio de Biotecnología, UNAN-Managua
Dra. Martha Lacayo Romero
Investigadora
Laboratorio de Biotecnología, UNAN-Managua
Lic. Daniel Tenorio Juárez
Investigador
Laboratorio de Biotecnología, UNAN-Managua
Palabras clave: ADN, extracción, kits comerciales, protocolos, recalcitrantes
RESUMEN
La obtención ácido desoxirribonucleico (ADN) purificado es un punto clave para el desarrollo de técnicas moleculares que permiten caracterizar una especie en estudio. Las matrices vegetales constituyen una de las muestras de mayor complejidad debido a los compuestos químicos que la forman y que muchas veces difieren su concentración en dependencia de la variedad en estudio; este es el caso de muestras recalcitrantes de variedades de cacao (Theobroma cacao) y café (Coffea arabica) donde la pérdida de ADN puede incluso ocurrir en la maceración de las muestras por fenolización del material. En el presente estudio se evaluaron tres kits comerciales disponibles mediante los cuales se obtienen excelentes recobros para variedades no recalcitrantes, así como también modificaciones realizadas a los protocolos tradicionales de Doyle & Doyle (1987) y Doyle & Doyle (1990). Los resultados obtenidos confirmaron el efecto de la variedad y la especie sobre el método de extracción, y de igual manera, que los métodos modificados pueden presentar mayor recobro y pureza que los kits comerciales para algunas especies vegetales.
INTRODUCCIÓN
El continuo crecimiento tecnológico acompañado de la necesidad de solucionar problemáticas prioritarias ha fomentado la implementación de técnicas moleculares como la reacción en cadena de la polimerasa (PCR) y la secuenciación. En el campo agrícola estas técnicas tienen aplicaciones que incluyen caracterizaciones moleculares, identificación de genes o mutaciones, entre otros, y en todos los casos la etapa inicial y fundamental es la extracción del ácido desoxirribonucleico (ADN) de plantas de interés industrial, ornamental y agroalimentario, en donde su utilidad está definida por la calidad y la cantidad obtenida.
De manera general, el aislamiento de ácidos nucleicos de alta calidad a partir del tejido de muchas plantas es notoriamente difícil (Hughes D.W. y Galau G., 1988). La lisis celular ineficiente o los altos niveles de actividad nucleasa han sido tratados de diferentes maneras, sin embargo en múltiples plantas superiores, la fase acuosa es afectada por metabolitos secundarios cuando son co-extractados en conjunto, especialmente en tejido maduro, interfiriendo con el aislamiento de ácidos nucleicos biológicamente activos (Baker, Rugh y Kamalay, 1990).
Las plantas recalcitrantes como el cacao (Theobroma cacao) y el café (Coffea arabica) son ricos en metabolitos secundarios como compuestos fenólicos al igual que en polisacáridos, estos compuestos presentes en el tejido vegetal también pueden inhibir la amplificación mediante PCR (Jobes, Hurley y Thien, 1995; Wilson, 1997) y dificultan la extracción de su ADN en la cantidad y calidad requerida. Adicionalmente, además de las características de las plantas analizadas se debe considerar su estado de madurez, la variedad o cultivar de procedencia y el tipo de tejido utilizado.
Actualmente existen diversos métodos de aislamiento de ADN que van desde químicos hasta comerciales, y a pesar que los kits de extracción de ADN promulgan remover la mayoría de los compuestos inhibitorios del tejido vegetal más eficientemente que los métodos “caseros” de aislamiento, la evidencia experimental continúa encontrando resultados no satisfactorios en las extracciones de ADN de matrices recalcitrantes o con características bioquímicas muy específicas (alto contenido de polisacáridos y polifenoles, entre otros), comprometiendo su calidad e incluso su aplicación a distintos análisis moleculares. Es debido a esta importancia que el protocolo de purificación es un criterio fundamental que debe ser considerado para los objetivos y el alcance de una investigación.
MATERIALES Y MÉTODOS
Colecta de las muestras
Las muestras de tejido foliar (hojas) utilizadas procedían de árboles de cacao con características acriolladas y variedades de café no clonal, con madurez intermedia, tamaño mediano y en buen estado fitosanitario. Las muestras de hoja y su peciolo fueron colectadas, desinfectadas y refrigeradas hasta su traslado al Laboratorio a fin de evitar su degradación (oxidación). En el Laboratorio las mismas fueron caracterizadas morfológicamente para corroborar su identidad y fueron almacenadas a -20°C debidamente identificadas hasta su procesamiento.
Procesamiento de las muestras
Se pesaron de 25-100 mg de tejido, y debido al alto índice de oxidación de las muestras se ensayaron dos diferentes métodos de homogenización: secado de la hoja a 45°C durante 16 horas combinado con la maceración con arena de cuarzo calcinada agregando cantidades variantes de polivinilpirrolidona (PVP), así como la utilización de nitrógeno líquido asegurando la maceración y conservación en frío de la muestra hasta su incubación. Se debe indicar que todos los micropilones utilizados fueron aclimatizados para evitar la pérdida de la muestra por aplicación de calor.
Metodologías de extracción de ADN vegetal
Se compararon 3 kits comerciales: Promega Wizard SV Genomic DNA Purification System (A2360), Roche High Pure PCR Template Preparation Kit (11 796 828 001), y Qiagen DNeasy Plant Mini Kit (69104). Adicionalmente se evaluaron los resultados obtenidos de la modificación a los protocolos químicos de extracción descritos por Doyle & Doyle (1987) y Doyle (1990) debido a que al implementar los protocolos originales no se obtuvo ADN de las muestras. Todos los ensayos con protocolos químicos fueron realizados por duplicado, con un control negativo. Se incluyeron muestras de pasto (Pennisetum purpureum) y neem (Azadirachta indica) como controles positivos de extracción en las pruebas donde se consideró pertinente debido al espectro del kit o el protocolo.
Las extracciones con los kits Promega (A2360), Roche (11 796 828 001) y Qiagen (69104) se realizaron de acuerdo al protocolo establecido por los fabricantes. La extracción realizada según el protocolo modificado de Doyle y Doyle (1987) partió de 100 mg de tejido foliar y 30 mg de arena de mar purificada, macerándose con 1000 μL de buffer de extracción de bromuro de hexadeciltrimetilamonio (CTAB, por sus siglas en inglés) 2X precalentado a 65°C. Una vez macerado se incubó a temperatura ambiente por 5 minutos y luego a -20°C por 12 minutos. Se adicionó 20 μL de RNasa A e incubó por 20 minutos a 37°C. Posteriormente se adicionó 10 μL de proteinasa K (13 mg/mL) incubándose nuevamente por 20 minutos a 60°C y 7 minutos en hielo. Se realizaron tres lavados con 600 μL de fenol:cloroformo:alcohol isoamílico (25:24:1). Luego, se adicionaron 50 μL de acetato de amonio 10M y se incubó a -20°C tras la adición de 500 μL isopropanol (4°C). Se decantó el sobrenadante posterior a la centrifugación y se adicionó 1 mL de etanol al 70% (-20°C) mezclándose por inversión, se colocaron en reposo por 5 minutos a temperatura ambiente y se procedió a centrifugar nuevamente para obtener el precipitado que fue secado por 40 minutos. Finalmente, el ADN se rehidrató con 200 μL de buffer TE 1X.
El protocolo Doyle (1990) con modificaciones de Shepherd & McLay (2011) y Tibbits, McMannus, Spokevicius, & Bossinger (2006) se implementó partiendo de 50 mg de tejido foliar macerado con nitrógeno líquido. Las modificaciones realizadas incluyen la homogeneización de los tiempos de agitación a máxima velocidad en Vortex por 1 minuto, además se adicionó de manera independiente 500 μL de buffer CTAB previamente calentado a 65°C, seguido por el β-mercaptoetanol utilizado al 99.9%, proteinasa K (13 mg/mL) y la mezcla de NaCl: BSA (5:1). Las muestras se incubaron por 20 h a 65°C mezclando en Vortex por 1 minuto durante y después de la incubación. Posteriormente, se adicionó isopropanol y se mezcló por inversión, se centrifugó e incubó a -20°C y en el primer paso de resuspensión en Tris-HCl 10 mM se almacenó a 4°C por 16 h. Cabe señalar que después de este período de reposo se añadió el acetato de sodio 2,4 M y etanol absoluto mezclándose por inversión seguido por centrifugación, lavado con etanol, secado del pellet y resuspensión en TE hasta almacenarse a 4°C.
Como prueba final de este protocolo modificado se utilizaron 50 mg de tejido foliar de una muestra de cacao (dos variedades) y café (un cultivar) adicionando una concentración menor de β-mercaptoetanol (10%) y se añadió un tercer lavado con cloroformo para obtener muestras más limpias, puesto que es necesaria la eliminación de metabolitos secundarios que pudieran interferir en la amplificación de las muestras utilizando la técnica de reacción en cadena de polimerasa (PCR, por sus siglas en inglés).
Evaluación de la calidad del ADN vegetal extraído
La calidad del ADN obtenido fue verificada mediante electroforesis en gel de agarosa con una concentración de 0.8% y tinción con bromuro de etidio. Para cargar las muestras en el gel estas se mezclaron con tinte de carga 6X (Thermo Fisher Scientific, R0611) y se utilizó un patrón marcador de peso molecular de 100 pb (Fisher Scientific, BP2573-100). Las geles se corrieron a 80V durante una hora y fueron visualizadas en un transiluminador ultravioleta modelo BBA (Fisher Scientific).
La cuantificación de la concentración de ADN de doble cadena (dsDNA, por sus siglas en inglés) se realizó mediante la determinación de su absorbancia a 260 nm en el espectrofotómetro Cary 50-BIO (Varian). La pureza de las muestras se determinó cuantificando la presencia de proteínas a 280 nm y calculando la relación A260/A280; asimismo se realizó la corrección de turbidez a 320 nm.
Finalmente, las muestras con los mejores resultados de calidad y pureza fueron amplificadas mediante la reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) utilizando marcadores moleculares microsatélites (SSR, por sus siglas en inglés) y verificadas en gel de agarosa al 1.8%. Durante el PCR se utilizaron volúmenes de reacción de 25 µL conteniendo 1 µL de ADN, 1X de Go Taq® Colorless Master Mix (M7133) y 1 µM de cada primer (Eurofins Genomics). El programa de amplificaciones consistió en la desnaturalización inicial (94°C) por 3 minutos (min.), seguido por 30 ciclos de 94°C por 30 segundos (s), 30 s a temperatura de alineamiento de cada primer y 45 s a 72°C, con una extensión final a 72°C por 3 min. Los productos de la amplificación fueron analizados por electroforesis en 1,8% de gel de agarosa Low EEO/Multipurpose (BP160-500) teñida con bromuro de etidio (10 mg/mL).
RESULTADOS Y DISCUSIÓN
Kit Promega Wizard SV Genomic DNA Purification System (A2360)
Se utilizaron 50 y 100 mg tanto de hojas frescas de cacao como pre-secadas al horno y maceradas con nitrógeno líquido, sin embargo no se visualizaron bandas en la gel de agarosa (0.8%), obteniendo resultados de baja calidad (figura 1).

Figura 1. Resultados de extracción con kit Promega (A2360) en gel de agarosa (0.8%). Mm: marcador de peso molecular de 100 pb, líneas 1 y 2: 50 mg de muestra pre-secada y macerada con arena, líneas 3 y 4: 100 mg de muestra pre-secada y macerada con arena, líneas 5 y 6: 50 mg de muestra macerada con nitrógeno líquidos, líneas 7 y 8: 100 mg de muestra macerada con nitrógeno líquido.
Kit Roche High Pure PCR Template Preparation (11 796 828 001)
La extracción se realizó utilizando desde 25 a 45 mg de muestras de cacao y gramínea (control) almacenada a -20°C y maceradas utilizando arena o nitrógeno líquido. Se obtuvieron resultados con calidad mínima para las muestras de gramínea y nulas para cacao (figura 2).

Figura 2. Resultados de extracción con kit Roche (11 796 828 001) en gel de agarosa (0.8%). 100 pb: marcador de peso molecular, línea 1: 25 mg de gramínea macerado con nitrógeno líquido, línea 2: 25 mg de gramínea pre-secada y macerada con arena, línea 3: 45 mg de gramínea macerado con nitrógeno líquido, línea 4: 45 mg de gramínea pre-secada y macerada con arena, líneas 5: 25 mg de cacao macerado con nitrógeno líquido, línea 6: 25 mg de cacao pre-secado y macerado con arena, línea 7: 45 mg de cacao macerado con nitrógeno líquido, línea 4: 45 mg de cacao pre-secado y macerado con arena.
Kit Qiagen DNeasy Plant Mini (69104)
La extracción se realizó a partir de 100 mg de tejido foliar de cacao provenientes de 3 árboles distintos maceradas con 30 mg de arena de mar purificada. Las muestras de ADN genómico extraído fueron analizadas tras su visualización en gel de agarosa 0.8% donde se encontró muy bajo recobro para las muestras 1 y 3, y mayor intensidad de banda en la muestra 2 (figura 3). Esto puede deberse a que existen diferencias en el contenido de compuestos que interfieren en el recobro del ADN en dependencia de la variedad en estudio.
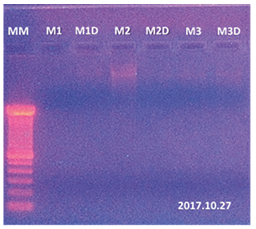
Figura 3. Resultados de extracción con kit Qiagen (69104) en gel de agarosa (0.8%). MM: marcador de peso molecular de 100 pb, líneas M1: 100 mg de muestra 1 de cacao, línea M1D: réplica de la muestra M1, línea M2: 100 mg de muestra 2 de cacao, línea M2D: réplica de la muestra M2, línea M3: 100 mg de muestra 3 de cacao, línea M3D: réplica de la muestra M3.
Protocolo Doyle y Doyle (1987) modificado
En este protocolo no se obtuvieron resultados positivos en la extracción, descartando este protocolo para las variedades en estudio (figura 4).

Figura 4. Resultados de extracción con protocolo Doyle y Doyle (1987) en gel de agarosa (0.8%). MM: marcador de peso molecular de 100 pb, líneas M1: 100 mg de muestra 1 de cacao, línea M1D: réplica de la muestra M1, línea M2: 100 mg de muestra 2 de cacao, línea M2D: réplica de la muestra M2, línea CN: control negativo del medio.
Protocolo Doyle (1990) con modificaciones de Shepherd & McLay (2011) y Tibbits, McMannus, Spokevicius, & Bossinger (2006)
La extracción de ADN genómico a partir de muestras de tejido foliar de cacao, gramínea y neem con su duplicado se realizó utilizando 50 mg de tejido foliar, (figura 5). En la gel se puede evidenciar la necesidad de lavados adicionales, lo cual constituyó la modificación final del protocolo (figura 6). Los resultados obtenidos de la modificación final del protocolo Doyle (1990) mostraron mejores recobros de ADN además de una calidad apropiada para la caracterización de las mismas.
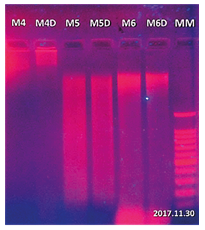
Figura 5. Resultados de extracción con protocolo Doyle (1990) en gel de agarosa (0.8%). MM: marcador de peso molecular de 100 pb, líneas M4: 50 mg de muestra de cacao, línea M4D: réplica de la muestra M4, línea M5: 50 mg de muestra de gramínea, línea M5D: réplica de la muestra M5, línea M6: 50 mg de muestra de neem, línea M6D: réplica de la muestra M6.
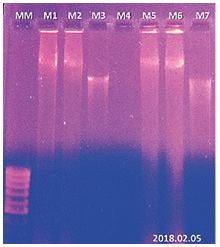
Figura 6. Resultados de última prueba con el protocolo modificado en gel de agarosa (0.8%). MM: marcador de peso molecular de 100 pb, línea M1: 50 mg de muestra de cacao, línea M2: réplica de la muestra M1, línea M3: 50 mg de muestra de café, línea M4: control negativo, línea M5: 50 mg de muestra de cacao, línea M6: réplica de la muestra M5, M7: 50 mg de muestra de café.
Cuantificación del dsDNA
La cantidad de ADN obtenida para las cinco metodologías de extracción es variable, tanto por método como por matriz (figura 7). En todos los casos las muestras de café presentaron un mejor recobro que las muestras de cacao. Con respecto a la metodología, en el protocolo Doyle (1990) modificado se determinó la mayor cantidad de ADN (0.99 mg/ml en café y 0.70 mg/ml en cacao), seguido por el kit Qiagen (0.53 mg/ml en café y 0.34 mg/ml en cacao) y el kit Promega (0.24 mg/ml en café y 0.17 mg/ml en cacao).
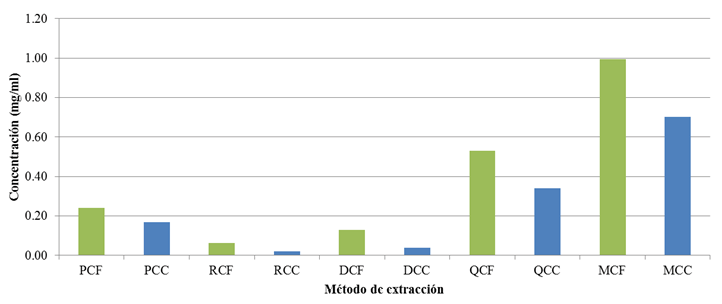
Figura 7. Concentración de ADN (mg/ml) a partir de cultivares de café (CF) y cacao (CC) obtenida mediante kits comerciales: Promega (P), Roche (R), Qiagen (Q) y métodos químicos: Doyle & Doyle (1987) (D) y Doyle (1990) con modificaciones de Shepherd & McLay (2011) y Tibbits, McMannus, Spokevicius, & Bossinger (2006) (M). PCF: Método Promega y muestras de café, PCC: Método Promega y muestras de cacao, RCF: Método Roche y muestras de café, RCC: Método Roche y muestras de cacao, DCF: Método Doyle & Doyle (1987) y muestras de café, DCC: Método Doyle & Doyle (1987) y muestras de cacao, QCF: Método Qiagen y muestras de café, QCC: Método Qiagen y muestras de cacao, MCF: Método Doyle (1990) modificado y muestras de café, MCC: Método Doyle (1990) modificado y muestras de cacao.
Pureza del ADN
De igual manera, la pureza de las muestras es variable en cuanto a metodología empleada, aunque debe señalarse que no presenta la misma variación observada en la cuantificación del ADN con respecto a la matriz (figura 8).
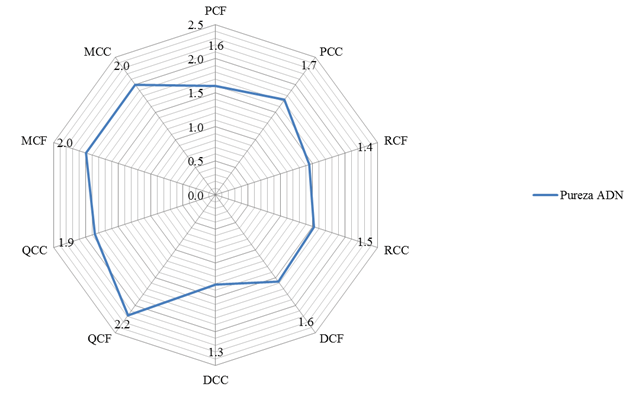
Figura 8. Pureza del ADN (mg/ml) de cultivares de café (CF) y cacao (CC) obtenida mediante kits comerciales: Promega (P), Roche (R), Qiagen (Q) y métodos químicos: Doyle & Doyle (1987) (D) y Doyle (1990) con modificaciones de Shepherd & McLay (2011) y Tibbits, McMannus, Spokevicius, & Bossinger (2006) (M).
En general, los resultados obtenidos tanto en la integridad de las muestras por electroforesis así como la concentración y pureza del ADN extraído coinciden con los inconvenientes descritos por Li, Su y Chen (2002) y Sharma et al. (2002), que describen la problemática relacionada con la co-extracción de polisacáridos y polifenoles que actúan como inhibidores durante la extracción del ADN, y que en dependencia de su concentración, pueden llegar a oxidar el ADN durante el proceso de lisis. Sharna et al. (2002) describe que incluso especies muy relacionadas pueden requerir diferentes protocolos de aislamiento de ADN, lo cual coincide con la experiencia previa a este ensayo donde se ha logrado recuperar material genético con el kit Qiagen, Promega y los métodos de Doyle & Doyle (1987) para ciertas variedades de cacao. Cabe señalar que las muestras de cacao, que reflejaron la menor concentración en todos los casos, son consideradas acriolladas por sus características productivas, morfológicas, además de su aroma y sabor.
De igual manera López P., López A. y Marulanda M. (2011), reporta en su estandarización de la extracción de ADN de Tabebuia rosea y Cordia alliodora, que mediante el kit Plant DNeasy mini de Qiagen no se lograron extraer y amplificar las muestras de T. rosea debido a que el kit requiere tejidos jóvenes o que contengan niveles bajos de polifenoles (Li et al. 2002), caso opuesto a las muestras seleccionadas que según lo indicado por Martínez (2010) y Arbeláez, Acevedo y Jaramillo (2011) presentan diversos metabolitos secundarios. Este tipo de muestras coincide con las características químicas del cacao y café.
Amplificación utilizando el ADN de mejor calidad
Posterior a la verificación de la calidad del ADN extraído, se seleccionó como mejor resultado la muestra de café obtenida mediante el protocolo modificado Doyle (1990) y se amplificó con 10 marcadores microsatélites (SSR) internacionalmente descritos para la caracterización molecular de café. Los resultados obtenidos fueron satisfactorios en cuanto a claridad y reproducibilidad en las bandas (figura 9).
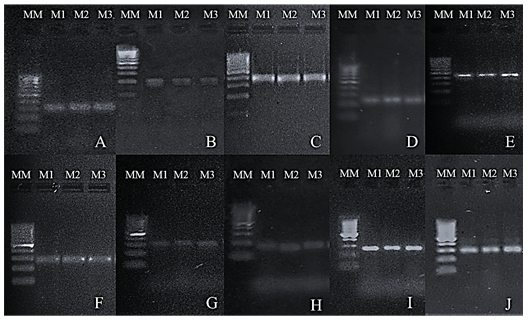
Figura 9. Productos de PCR obtenidos de la amplificación del ADN de café extraído mediante el protocolo modificado de Doyle (1990) utilizando 10 pares de marcadores en gel de agarosa Low EEO/Multipurpose (1.8%). MM: marcador de peso molecular de 100 pb. M1, M2 y M3: réplicas de las muestras. (A) Amplicones con el marcador CarM096. (B) Amplicones con el marcador CarM101. (C) Amplicones con el marcador CarM049. (D) Amplicones con el marcador CarM069. (E) Amplicones con el marcador CarM092. (F) Amplicones con el marcador CarM105. (G) Amplicones con el marcador M20. (H) Amplicones con el marcador M24. (I) Amplicones con el marcador CarM051. (J) Amplicones con el marcador CarM050.
CONCLUSIONES
La calidad y cantidad del ADN extraído se ve influida por la metodología utilizada así como por la naturaleza de la matriz en estudio. De los métodos comerciales utilizados, los mejores resultados en cuanto a cantidad y pureza se obtuvieron mediante la extracción con el kit Qiagen, aunque debe señalarse que los resultados fueron de bajo recobro. En cuanto a los protocolos evaluados, Doyle (1990) con modificaciones de Shepherd & McLay (2011) y Tibbits, McMannus, Spokevicius, & Bossinger (2006) presentó los mejores recobros y pureza, incluso superior que el kit comercial. Cabe señalar que este comportamiento no puede ser generalizado a todas las variedades vegetales, incluso de una misma especie, debido a que mediante los kits Promega y Qiagen, al igual que con la metodología Doyle y Doyle (1987), se han obtenido buenos recobros de ADN para otras variedades de cacao y café evidenciando claramente la influencia de la variedad de las especies de la cual se obtiene el tejido a analizar.
AGRADECIMIENTOS
Este estudio se realizó con apoyo del financiamiento recibido por el Fondo para Proyectos de Investigación (FPI) de UNAN-Managua.
REFERENCIAS BIBLIOGRÁFICAS
Alejos, L., Aragón, M., & Cornejo, A. (2008). Extracción y purificación de ADN. México: UNAM.
Arbeláez, J., Acevedo, J. y Jaramillo, R. (2011). Metabolitos secundarios en el guayacán amarillo y en el guayacán rosado. Scientia et Technica 47:297- 301.
Baker S.S., Rugh C.L. y Kamalay J.C. (1990). RNA and DNA isolation from recalcitrant plant tissues. Biotechniques; 9(3):268-72.
Doyle, J. (1990). DNA PROTOCOLS FOR PLANTS. NATO ASY Series, 283-284.
Doyle, J., & Doyle, J. (1987). A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochemical bulletin, 19:11–15.
Hughes D.W. y Galau G. (1988). Preparation of RNA from cotton leaves and pollen. Plant Molecular Biology Rep. 6:253-257.
Jobes D. V., Hurley D. L. y Thien L.B. (1995). Plant DNA isolation: A method to effi ciently remove polyphenolics, polysaccharides, and RNA. Taxon 44: 379 – 386.
Li, Y. X., Su, Z. X. y Chen, F. (2002). Rapid Extraction of Genomic DNA From Leaves and Bracts of Dove Tree (Davidia involucrata). Plant Molecular Biology Reporter 20(2):185.
Lopez Mora, P., Lopez Gutierrez, A., & Marulanda-Angel, M. (2011). Estandarización de la extracción de adn genómico en Tabebuia rosea (Bertol.) DC. y Cordia alliodora (Ruiz y Pav.) Okén. Temas Agrarios, 16(2), 28-41.
Martínez, A.J. (2010). Principios activos en el Guayacán Amarillo (Tabebuia chrysantha Jacq. Nicholson, Bignoniaceae) y en el Guayacán Rosado (Tabebuia rosea Bertol. DC). Proyecto de Grado, Universidad del Quindío, Armenia - Colombia.
McLay, T. (4 de Septiembre de 2017). Hight quality DNA extraction protocol from recalcitrant plant tissues. Obtenido de protocols.io.
QIAGEN. (2006). Protocolo: Purification of total DNA from Plant Tissue (Miniprotocol). Estados Unidos: QIAGEN Group.
QIAGEN. (2011). DNeasy® Plant Mini Kit . Estados Unidos: QIAGEN Group.
Sharma, A.D., Gill, P.K. y Singhdna, P. (2002). Isolation From Dry and Fresh Samples of Polysaccharide-Rich Plants. Plant Molecular Biology Reporter 20(4):415.
Shepherd, L., & McLay, T. (2011). Two micro-scale protocols for the isolation of DNA from polysaccharide-rich plant tissue. Journal of Plant Research, 311-314.
Tibbits, J., McMannus, L., Spokevicius, A., & Bossinger, G. (2006). A rapid method for tissue collection and high-throughput isolation of genomic DNA from mature trees. Plant Molecular Biology Reporter, 81-91.
Wilson, I. G. (1997). Inhibition and facilitation of nucleic acid amplification. Applied and Environmental Microbiology 63: 3741 – 3751.